class: center, middle, inverse, title-slide # Analisi Esplorativa ## Analisi delle componenti principali: applicazioni ### Aldo Solari --- # Outline * `USArrests` data, discusso in [An Introduction to Statistical Learning](https://hastie.su.domains/ISLR2/ISLRv2_website.pdf), Sezione 12.2 e 12.5.1 * Valori mancanti e completamento della matrice, discusso in [An Introduction to Statistical Learning](https://hastie.su.domains/ISLR2/ISLRv2_website.pdf), Sezione 12.3 e 12.5.2 * `heptathlon` data, discusso in An introduction to Applied Multivariate Analysis with R, Sezione 3.10.2 --- # `USArrests` Per ciascuno dei `\(n=50\)` stati negli Stati Uniti, il data set contiene il numero degli arresti per 100.000 residenti per ciascuno dei tre reati: * aggressione (`Assault`) * omicidio (`Murder`) * stupro (`Rape`) E' inoltre presente la variabile `UrbanPop`, che indica la percentuale della popolazione in ogni stato che vive nelle aree urbane --- ```r X <- USArrests n <- nrow(X) p <- ncol(X) states <- row.names(X) names(X) ``` ``` [1] "Murder" "Assault" "UrbanPop" "Rape" ``` ```r head(X) ``` ``` Murder Assault UrbanPop Rape Alabama 13.2 236 58 21.2 Alaska 10.0 263 48 44.5 Arizona 8.1 294 80 31.0 Arkansas 8.8 190 50 19.5 California 9.0 276 91 40.6 Colorado 7.9 204 78 38.7 ``` --- ```r apply(X, 2, mean) ``` ``` Murder Assault UrbanPop Rape 7.788 170.760 65.540 21.232 ``` ```r apply(X, 2, var) * (n-1)/n ``` ``` Murder Assault UrbanPop Rape 18.59106 6806.26240 205.32840 85.97458 ``` Se non standardizziamo le variabili prima di eseguire la PCA, le componenti principali saranno influenzate dalla variabile `Assault` che presenta variabilità molto elevata. Per questi dati è importante standardizzare i dati prima di eseguire PCA. --- ```r devstd <- sqrt( apply(X, 2, var) * (n-1)/n ) Z <- scale(X, center=TRUE, scale = devstd) R <- cor(X) R ``` ``` Murder Assault UrbanPop Rape Murder 1.00000000 0.8018733 0.06957262 0.5635788 Assault 0.80187331 1.0000000 0.25887170 0.6652412 UrbanPop 0.06957262 0.2588717 1.00000000 0.4113412 Rape 0.56357883 0.6652412 0.41134124 1.0000000 ``` --- ```r eigenR <- eigen(R) Lambda <- diag(eigenR$values) Lambda ``` ``` [,1] [,2] [,3] [,4] [1,] 2.480242 0.0000000 0.0000000 0.0000000 [2,] 0.000000 0.9897652 0.0000000 0.0000000 [3,] 0.000000 0.0000000 0.3565632 0.0000000 [4,] 0.000000 0.0000000 0.0000000 0.1734301 ``` ```r # pesi V <- eigenR$vectors V ``` ``` [,1] [,2] [,3] [,4] [1,] -0.5358995 0.4181809 -0.3412327 0.64922780 [2,] -0.5831836 0.1879856 -0.2681484 -0.74340748 [3,] -0.2781909 -0.8728062 -0.3780158 0.13387773 [4,] -0.5434321 -0.1673186 0.8177779 0.08902432 ``` ```r # punteggi Y <- Z %*% V head(Y) ``` ``` [,1] [,2] [,3] [,4] Alabama -0.9855659 1.1333924 -0.44426879 0.156267145 Alaska -1.9501378 1.0732133 2.04000333 -0.438583440 Arizona -1.7631635 -0.7459568 0.05478082 -0.834652924 Arkansas 0.1414203 1.1197968 0.11457369 -0.182810896 California -2.5239801 -1.5429340 0.59855680 -0.341996478 Colorado -1.5145629 -0.9875551 1.09500699 0.001464887 ``` --- # `princomp()` ```r # cor = TRUE per PCA su dati standardizzati ACP <- princomp(X, cor=TRUE) names(ACP) ``` ``` [1] "sdev" "loadings" "center" "scale" "n.obs" "scores" "call" ``` --- ```r # Standard deviation coincide con sqrt(diag(Lambda)) summary(ACP) ``` ``` Importance of components: Comp.1 Comp.2 Comp.3 Comp.4 Standard deviation 1.5748783 0.9948694 0.5971291 0.41644938 Proportion of Variance 0.6200604 0.2474413 0.0891408 0.04335752 Cumulative Proportion 0.6200604 0.8675017 0.9566425 1.00000000 ``` ```r # pesi ACP$loadings[,] ``` ``` Comp.1 Comp.2 Comp.3 Comp.4 Murder 0.5358995 0.4181809 0.3412327 0.64922780 Assault 0.5831836 0.1879856 0.2681484 -0.74340748 UrbanPop 0.2781909 -0.8728062 0.3780158 0.13387773 Rape 0.5434321 -0.1673186 -0.8177779 0.08902432 ``` ```r # punteggi ACP$scores ``` ``` Comp.1 Comp.2 Comp.3 Comp.4 Alabama 0.98556588 1.13339238 0.44426879 0.156267145 Alaska 1.95013775 1.07321326 -2.04000333 -0.438583440 Arizona 1.76316354 -0.74595678 -0.05478082 -0.834652924 Arkansas -0.14142029 1.11979678 -0.11457369 -0.182810896 California 2.52398013 -1.54293399 -0.59855680 -0.341996478 Colorado 1.51456286 -0.98755509 -1.09500699 0.001464887 Connecticut -1.35864746 -1.08892789 0.64325757 -0.118469414 Delaware 0.04770931 -0.32535892 0.71863294 -0.881977637 Florida 3.01304227 0.03922851 0.57682949 -0.096284752 Georgia 1.63928304 1.27894240 0.34246008 1.076796812 Hawaii -0.91265715 -1.57046001 -0.05078189 0.902806864 Idaho -1.63979985 0.21097292 -0.25980134 -0.499104101 Illinois 1.37891072 -0.68184119 0.67749564 -0.122021292 Indiana -0.50546136 -0.15156254 -0.22805484 0.424665700 Iowa -2.25364607 -0.10405407 -0.16456432 0.017555916 Kansas -0.79688112 -0.27016470 -0.02555331 0.206496428 Kentucky -0.75085907 0.95844029 0.02836942 0.670556671 Louisiana 1.56481798 0.87105466 0.78348036 0.454728038 Maine -2.39682949 0.37639158 0.06568239 -0.330459817 Maryland 1.76336939 0.42765519 0.15725013 -0.559069521 Massachusetts -0.48616629 -1.47449650 0.60949748 -0.179598963 Michigan 2.10844115 -0.15539682 -0.38486858 0.102372019 Minnesota -1.69268181 -0.63226125 -0.15307043 0.067316885 Mississippi 0.99649446 2.39379599 0.74080840 0.215508013 Missouri 0.69678733 -0.26335479 -0.37744383 0.225824461 Montana -1.18545191 0.53687437 -0.24688932 0.123742227 Nebraska -1.26563654 -0.19395373 -0.17557391 0.015892888 Nevada 2.87439454 -0.77560020 -1.16338049 0.314515476 New Hampshire -2.38391541 -0.01808229 -0.03685539 -0.033137338 New Jersey 0.18156611 -1.44950571 0.76445355 0.243382700 New Mexico 1.98002375 0.14284878 -0.18369218 -0.339533597 New York 1.68257738 -0.82318414 0.64307509 -0.013484369 North Carolina 1.12337861 2.22800338 0.86357179 -0.954381667 North Dakota -2.99222562 0.59911882 -0.30127728 -0.253987327 Ohio -0.22596542 -0.74223824 0.03113912 0.473915911 Oklahoma -0.31178286 -0.28785421 0.01530979 0.010332321 Oregon 0.05912208 -0.54141145 -0.93983298 -0.237780688 Pennsylvania -0.88841582 -0.57110035 0.40062871 0.359061124 Rhode Island -0.86377206 -1.49197842 1.36994570 -0.613569430 South Carolina 1.32072380 1.93340466 0.30053779 -0.131466685 South Dakota -1.98777484 0.82334324 -0.38929333 -0.109571764 Tennessee 0.99974168 0.86025130 -0.18808295 0.652864291 Texas 1.35513821 -0.41248082 0.49206886 0.643195491 Utah -0.55056526 -1.47150461 -0.29372804 -0.082314047 Vermont -2.80141174 1.40228806 -0.84126309 -0.144889914 Virginia -0.09633491 0.19973529 -0.01171254 0.211370813 Washington -0.21690338 -0.97012418 -0.62487094 -0.220847793 West Virginia -2.10858541 1.42484670 -0.10477467 0.131908831 Wisconsin -2.07971417 -0.61126862 0.13886500 0.184103743 Wyoming -0.62942666 0.32101297 0.24065923 -0.166651801 ``` --- # `prcomp()` ```r ACP2 <- prcomp(X, scale=TRUE) names(ACP2) ``` ``` [1] "sdev" "rotation" "center" "scale" "x" ``` ```r # le deviazioni standard sono calcolate con il denominatore n-1 ACP2$scale ``` ``` Murder Assault UrbanPop Rape 4.355510 83.337661 14.474763 9.366385 ``` ```r devstd * sqrt(n/(n-1)) ``` ``` Murder Assault UrbanPop Rape 4.355510 83.337661 14.474763 9.366385 ``` Inoltre, l'help della funzione specifica: *Unlike princomp, variances are computed with the usual divisor N - 1*. --- ```r summary(ACP2) ``` ``` Importance of components: PC1 PC2 PC3 PC4 Standard deviation 1.5749 0.9949 0.59713 0.41645 Proportion of Variance 0.6201 0.2474 0.08914 0.04336 Cumulative Proportion 0.6201 0.8675 0.95664 1.00000 ``` ```r # pesi ACP2$rotation[,] ``` ``` PC1 PC2 PC3 PC4 Murder -0.5358995 0.4181809 -0.3412327 0.64922780 Assault -0.5831836 0.1879856 -0.2681484 -0.74340748 UrbanPop -0.2781909 -0.8728062 -0.3780158 0.13387773 Rape -0.5434321 -0.1673186 0.8177779 0.08902432 ``` ```r # punteggi ACP2$x ``` ``` PC1 PC2 PC3 PC4 Alabama -0.97566045 1.12200121 -0.43980366 0.154696581 Alaska -1.93053788 1.06242692 2.01950027 -0.434175454 Arizona -1.74544285 -0.73845954 0.05423025 -0.826264240 Arkansas 0.13999894 1.10854226 0.11342217 -0.180973554 California -2.49861285 -1.52742672 0.59254100 -0.338559240 Colorado -1.49934074 -0.97762966 1.08400162 0.001450164 Connecticut 1.34499236 -1.07798362 -0.63679250 -0.117278736 Delaware -0.04722981 -0.32208890 -0.71141032 -0.873113315 Florida -2.98275967 0.03883425 -0.57103206 -0.095317042 Georgia -1.62280742 1.26608838 -0.33901818 1.065974459 Hawaii 0.90348448 -1.55467609 0.05027151 0.893733198 Idaho 1.62331903 0.20885253 0.25719021 -0.494087852 Illinois -1.36505197 -0.67498834 -0.67068647 -0.120794916 Indiana 0.50038122 -0.15003926 0.22576277 0.420397595 Iowa 2.23099579 -0.10300828 0.16291036 0.017379470 Kansas 0.78887206 -0.26744941 0.02529648 0.204421034 Kentucky 0.74331256 0.94880748 -0.02808429 0.663817237 Louisiana -1.54909076 0.86230011 -0.77560598 0.450157791 Maine 2.37274014 0.37260865 -0.06502225 -0.327138529 Maryland -1.74564663 0.42335704 -0.15566968 -0.553450589 Massachusetts 0.48128007 -1.45967706 -0.60337172 -0.177793902 Michigan -2.08725025 -0.15383500 0.38100046 0.101343128 Minnesota 1.67566951 -0.62590670 0.15153200 0.066640316 Mississippi -0.98647919 2.36973712 -0.73336290 0.213342049 Missouri -0.68978426 -0.26070794 0.37365033 0.223554811 Montana 1.17353751 0.53147851 0.24440796 0.122498555 Nebraska 1.25291625 -0.19200440 0.17380930 0.015733156 Nevada -2.84550542 -0.76780502 1.15168793 0.311354436 New Hampshire 2.35995585 -0.01790055 0.03648498 -0.032804291 New Jersey -0.17974128 -1.43493745 -0.75677041 0.240936580 New Mexico -1.96012351 0.14141308 0.18184598 -0.336121113 New York -1.66566662 -0.81491072 -0.63661186 -0.013348844 North Carolina -1.11208808 2.20561081 -0.85489245 -0.944789648 North Dakota 2.96215223 0.59309738 0.29824930 -0.251434626 Ohio 0.22369436 -0.73477837 -0.03082616 0.469152817 Oklahoma 0.30864928 -0.28496113 -0.01515592 0.010228476 Oregon -0.05852787 -0.53596999 0.93038718 -0.235390872 Pennsylvania 0.87948680 -0.56536050 -0.39660218 0.355452378 Rhode Island 0.85509072 -1.47698328 -1.35617705 -0.607402746 South Carolina -1.30744986 1.91397297 -0.29751723 -0.130145378 South Dakota 1.96779669 0.81506822 0.38538073 -0.108470512 Tennessee -0.98969377 0.85160534 0.18619262 0.646302674 Texas -1.34151838 -0.40833518 -0.48712332 0.636731051 Utah 0.54503180 -1.45671524 0.29077592 -0.081486749 Vermont 2.77325613 1.38819435 0.83280797 -0.143433697 Virginia 0.09536670 0.19772785 0.01159482 0.209246429 Washington 0.21472339 -0.96037394 0.61859067 -0.218628161 West Virginia 2.08739306 1.41052627 0.10372163 0.130583080 Wisconsin 2.05881199 -0.60512507 -0.13746933 0.182253407 Wyoming 0.62310061 0.31778662 -0.23824049 -0.164976866 ``` --- ```r ACP3 <- prcomp(X, scale=devstd) # Standard deviation coincide con sqrt(diag(Lambda) * n/(n-1) ) summary(ACP3) ``` ``` Importance of components: PC1 PC2 PC3 PC4 Standard deviation 1.5909 1.0050 0.60319 0.42068 Proportion of Variance 0.6201 0.2474 0.08914 0.04336 Cumulative Proportion 0.6201 0.8675 0.95664 1.00000 ``` ```r # pesi ACP3$rotation[,] ``` ``` PC1 PC2 PC3 PC4 Murder -0.5358995 0.4181809 -0.3412327 0.64922780 Assault -0.5831836 0.1879856 -0.2681484 -0.74340748 UrbanPop -0.2781909 -0.8728062 -0.3780158 0.13387773 Rape -0.5434321 -0.1673186 0.8177779 0.08902432 ``` ```r # punteggi ACP3$x ``` ``` PC1 PC2 PC3 PC4 Alabama -0.98556588 1.13339238 -0.44426879 0.156267145 Alaska -1.95013775 1.07321326 2.04000333 -0.438583440 Arizona -1.76316354 -0.74595678 0.05478082 -0.834652924 Arkansas 0.14142029 1.11979678 0.11457369 -0.182810896 California -2.52398013 -1.54293399 0.59855680 -0.341996478 Colorado -1.51456286 -0.98755509 1.09500699 0.001464887 Connecticut 1.35864746 -1.08892789 -0.64325757 -0.118469414 Delaware -0.04770931 -0.32535892 -0.71863294 -0.881977637 Florida -3.01304227 0.03922851 -0.57682949 -0.096284752 Georgia -1.63928304 1.27894240 -0.34246008 1.076796812 Hawaii 0.91265715 -1.57046001 0.05078189 0.902806864 Idaho 1.63979985 0.21097292 0.25980134 -0.499104101 Illinois -1.37891072 -0.68184119 -0.67749564 -0.122021292 Indiana 0.50546136 -0.15156254 0.22805484 0.424665700 Iowa 2.25364607 -0.10405407 0.16456432 0.017555916 Kansas 0.79688112 -0.27016470 0.02555331 0.206496428 Kentucky 0.75085907 0.95844029 -0.02836942 0.670556671 Louisiana -1.56481798 0.87105466 -0.78348036 0.454728038 Maine 2.39682949 0.37639158 -0.06568239 -0.330459817 Maryland -1.76336939 0.42765519 -0.15725013 -0.559069521 Massachusetts 0.48616629 -1.47449650 -0.60949748 -0.179598963 Michigan -2.10844115 -0.15539682 0.38486858 0.102372019 Minnesota 1.69268181 -0.63226125 0.15307043 0.067316885 Mississippi -0.99649446 2.39379599 -0.74080840 0.215508013 Missouri -0.69678733 -0.26335479 0.37744383 0.225824461 Montana 1.18545191 0.53687437 0.24688932 0.123742227 Nebraska 1.26563654 -0.19395373 0.17557391 0.015892888 Nevada -2.87439454 -0.77560020 1.16338049 0.314515476 New Hampshire 2.38391541 -0.01808229 0.03685539 -0.033137338 New Jersey -0.18156611 -1.44950571 -0.76445355 0.243382700 New Mexico -1.98002375 0.14284878 0.18369218 -0.339533597 New York -1.68257738 -0.82318414 -0.64307509 -0.013484369 North Carolina -1.12337861 2.22800338 -0.86357179 -0.954381667 North Dakota 2.99222562 0.59911882 0.30127728 -0.253987327 Ohio 0.22596542 -0.74223824 -0.03113912 0.473915911 Oklahoma 0.31178286 -0.28785421 -0.01530979 0.010332321 Oregon -0.05912208 -0.54141145 0.93983298 -0.237780688 Pennsylvania 0.88841582 -0.57110035 -0.40062871 0.359061124 Rhode Island 0.86377206 -1.49197842 -1.36994570 -0.613569430 South Carolina -1.32072380 1.93340466 -0.30053779 -0.131466685 South Dakota 1.98777484 0.82334324 0.38929333 -0.109571764 Tennessee -0.99974168 0.86025130 0.18808295 0.652864291 Texas -1.35513821 -0.41248082 -0.49206886 0.643195491 Utah 0.55056526 -1.47150461 0.29372804 -0.082314047 Vermont 2.80141174 1.40228806 0.84126309 -0.144889914 Virginia 0.09633491 0.19973529 0.01171254 0.211370813 Washington 0.21690338 -0.97012418 0.62487094 -0.220847793 West Virginia 2.10858541 1.42484670 0.10477467 0.131908831 Wisconsin 2.07971417 -0.61126862 -0.13886500 0.184103743 Wyoming 0.62942666 0.32101297 -0.24065923 -0.166651801 ``` --- # SVD Si consideri la matrice dati standardizzati `\(Z\)`. Per il teorema di decomposizione spettrale, `\(S^Z = \frac{1}{n}Z'Z = R = V\Lambda V'\)`. Sia `\(Z = U\Delta V'\)` la decomposizione in valori singolari (SVD) di `\(Z\)`. Possiamo scrivere `$$R = \frac{1}{n}Z'Z = \frac{1}{n}(U\Delta V')'(U\Delta V') = \frac{1}{n}V \Delta U'U \Delta V' = V (\frac{1}{n}\Delta^2) V' = V \Lambda V'$$` quindi `\(\lambda_i = d^2_i/n\)` e possiamo ottenere la matrice dei pesi `\(V\)` da SVD di `\(Z\)`. Infine possiamo ottenere la matrice dei punteggi come `$$Y = ZV = U\Delta V' V = U\Delta$$` --- ```r DVS = svd(Z) U = DVS$u Delta = diag(DVS$d) diag(Delta)/sqrt(n) ``` ``` [1] 1.5748783 0.9948694 0.5971291 0.4164494 ``` ```r # pesi DVS$v ``` ``` [,1] [,2] [,3] [,4] [1,] -0.5358995 0.4181809 -0.3412327 0.64922780 [2,] -0.5831836 0.1879856 -0.2681484 -0.74340748 [3,] -0.2781909 -0.8728062 -0.3780158 0.13387773 [4,] -0.5434321 -0.1673186 0.8177779 0.08902432 ``` ```r # punteggi U %*% Delta ``` ``` [,1] [,2] [,3] [,4] [1,] -0.98556588 1.13339238 -0.44426879 0.156267145 [2,] -1.95013775 1.07321326 2.04000333 -0.438583440 [3,] -1.76316354 -0.74595678 0.05478082 -0.834652924 [4,] 0.14142029 1.11979678 0.11457369 -0.182810896 [5,] -2.52398013 -1.54293399 0.59855680 -0.341996478 [6,] -1.51456286 -0.98755509 1.09500699 0.001464887 [7,] 1.35864746 -1.08892789 -0.64325757 -0.118469414 [8,] -0.04770931 -0.32535892 -0.71863294 -0.881977637 [9,] -3.01304227 0.03922851 -0.57682949 -0.096284752 [10,] -1.63928304 1.27894240 -0.34246008 1.076796812 [11,] 0.91265715 -1.57046001 0.05078189 0.902806864 [12,] 1.63979985 0.21097292 0.25980134 -0.499104101 [13,] -1.37891072 -0.68184119 -0.67749564 -0.122021292 [14,] 0.50546136 -0.15156254 0.22805484 0.424665700 [15,] 2.25364607 -0.10405407 0.16456432 0.017555916 [16,] 0.79688112 -0.27016470 0.02555331 0.206496428 [17,] 0.75085907 0.95844029 -0.02836942 0.670556671 [18,] -1.56481798 0.87105466 -0.78348036 0.454728038 [19,] 2.39682949 0.37639158 -0.06568239 -0.330459817 [20,] -1.76336939 0.42765519 -0.15725013 -0.559069521 [21,] 0.48616629 -1.47449650 -0.60949748 -0.179598963 [22,] -2.10844115 -0.15539682 0.38486858 0.102372019 [23,] 1.69268181 -0.63226125 0.15307043 0.067316885 [24,] -0.99649446 2.39379599 -0.74080840 0.215508013 [25,] -0.69678733 -0.26335479 0.37744383 0.225824461 [26,] 1.18545191 0.53687437 0.24688932 0.123742227 [27,] 1.26563654 -0.19395373 0.17557391 0.015892888 [28,] -2.87439454 -0.77560020 1.16338049 0.314515476 [29,] 2.38391541 -0.01808229 0.03685539 -0.033137338 [30,] -0.18156611 -1.44950571 -0.76445355 0.243382700 [31,] -1.98002375 0.14284878 0.18369218 -0.339533597 [32,] -1.68257738 -0.82318414 -0.64307509 -0.013484369 [33,] -1.12337861 2.22800338 -0.86357179 -0.954381667 [34,] 2.99222562 0.59911882 0.30127728 -0.253987327 [35,] 0.22596542 -0.74223824 -0.03113912 0.473915911 [36,] 0.31178286 -0.28785421 -0.01530979 0.010332321 [37,] -0.05912208 -0.54141145 0.93983298 -0.237780688 [38,] 0.88841582 -0.57110035 -0.40062871 0.359061124 [39,] 0.86377206 -1.49197842 -1.36994570 -0.613569430 [40,] -1.32072380 1.93340466 -0.30053779 -0.131466685 [41,] 1.98777484 0.82334324 0.38929333 -0.109571764 [42,] -0.99974168 0.86025130 0.18808295 0.652864291 [43,] -1.35513821 -0.41248082 -0.49206886 0.643195491 [44,] 0.55056526 -1.47150461 0.29372804 -0.082314047 [45,] 2.80141174 1.40228806 0.84126309 -0.144889914 [46,] 0.09633491 0.19973529 0.01171254 0.211370813 [47,] 0.21690338 -0.97012418 0.62487094 -0.220847793 [48,] 2.10858541 1.42484670 0.10477467 0.131908831 [49,] 2.07971417 -0.61126862 -0.13886500 0.184103743 [50,] 0.62942666 0.32101297 -0.24065923 -0.166651801 ``` --- ```r biplot(ACP , scale = 0) ``` 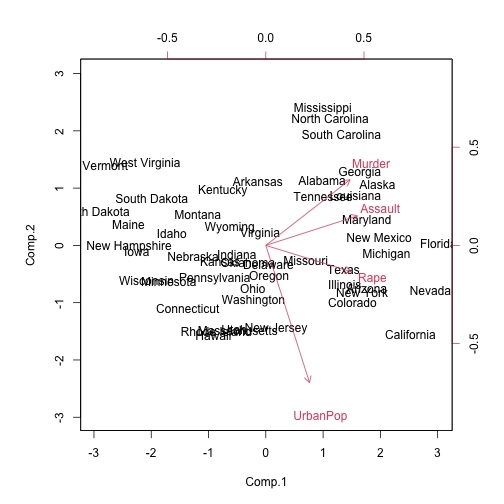<!-- --> --- Possiamo notare che i pesi della prima componente sono approssimativamente uguali per `Assault`, `Murder` e `Rape`, ma inferiore per `UrbanPop`. Questa componente corrisponde approssimativamente a una misura complessiva di tassi di reati gravi. Il secondo vettore dei pesi pone la maggior parte del suo peso su `UrbanPop` e molto meno peso sulle altre tre variabili. Questa componente corrisponde grosso modo al livello di urbanizzazione dello stato. Nella figura prececente, vediamo che le variabili relative alla criminalità si trovano vicine, mentre la variabile UrbanPop è lontana. Ciò indica che le variabili relative alla criminalità sono correlate tra loro, e che la variabile `UrbanPop` è meno correlata con le altre. La figura suggerisce che gli stati con punteggi positivi sulla prima componente, come California, Nevada e Florida, hanno alti tassi di criminalità, mentre Stati come il North Dakota, con punteggi negativi sulla prima componente, hanno bassi tassi di criminalità. La California ha anche un punteggio basso sulla secondo componente, indica un alto livello di urbanizzazione, mentre è vero il contrario per gli stati come il Mississippi. Stati vicini allo zero su entrambe le componenti, come Indiana, hanno livelli approssimativamente medi sia di criminalità che di urbanizzazione. --- # Correlazione tra componenti principali e variabili Qual è la variabile (`Assault`, `Murder`, `Rape`, `UrbanPop`) più correlata (in valore assoluto) con la prima componente principale? ```r k = 1 # prima componente principale sapply(1:4, function(j) V[j,k]*sqrt(diag(Lambda)[k]) ) ``` ``` [1] -0.8439764 -0.9184432 -0.4381168 -0.8558394 ``` ```r # corrisponde a cor(Y[,k], X) ``` ``` Murder Assault UrbanPop Rape [1,] -0.8439764 -0.9184432 -0.4381168 -0.8558394 ``` --- # Teorema di Eckart-Young La soluzione di `$$\min_{A \in \mathbb{R}^{n\times q}, B \in \mathbb{R}^{p\times q} }\Big\{ \sum_{i=1}^{n}\sum_{j=1}^{p}(z_{ij} - \sum_{k=1}^{q} a_{ik} b_{jk} )^2 \Big\}$$` è data da `\(A = Y_q\)` e `\(B = V_q\)`, quindi `\(z_{ij} \approx \sum_{k=1}^{q} y_{ik} v_{jk} = \{Y_q V_q'\}_{ij}\)` ```r q = 2 Yq = Y[,1:q, drop=FALSE] Vq = V[,1:q, drop=FALSE] Zapp = Yq %*% t(Vq) sum(c(Z - Zapp)^2) ``` ``` [1] 26.49966 ``` ```r # corrisponde a n * sum(diag(Lambda)[(q+1):p]) ``` ``` [1] 26.49966 ``` --- # Analisi sui dati non standardizzati .pull-left[ ```r plot(UrbanPop ~ Assault, X, asp=1) ``` <!-- --> ] .pull-right[ ```r biplot(princomp(X), scale=0) ``` 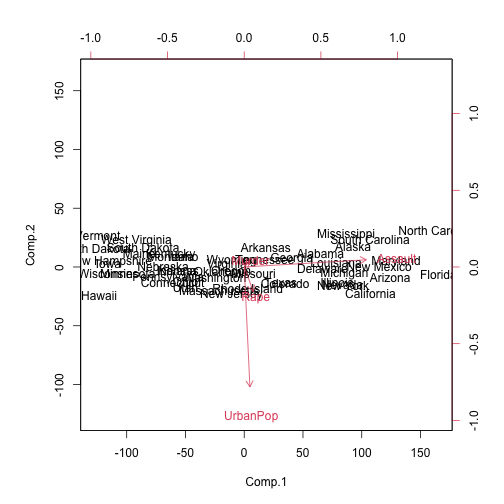<!-- --> ] --- # Valori mancanti e completamento della matrice Spesso i set di dati presentano valori mancanti. Per esempio, supponiamo di voler analizzare i dati di `USArrests` e scoprire che 20 dei 200 valori sono stati rimossi casualmente e contrassegnati come mancanti. ```r nomit <- 20 set.seed(123) ina <- sample(seq(n), nomit) inb <- sample(1:p, nomit, replace = TRUE) Zna <- Z index.na <- cbind(ina, inb) Zna[index.na] <- NA ``` Sfortunatamente, l'analisi delle componenti principali non prevede valori mancanti. Come dobbiamo procedere? --- Possiamo risolvere `$$\min_{A \in \mathbb{R}^{n\times q}, B \in \mathbb{R}^{p\times q} }\Big\{ \sum_{(i,j) \in \mathcal{O}}(z_{ij} - \sum_{k=1}^{q} a_{ik} b_{jk} )^2 \Big\}$$` dove `\(\mathcal{O}\)` è l'insieme delle coppie di indici `\((i,j)\)` osservati. Una volta trovate le soluzioni `\(A^*\)` e `\(B^*\)`, possiamo * sostituire le osservazioni mancanti `\(z_{ij}\)` con `\(z^*_{ij} = \sum_{k=1}^{q}a^*_{ik} b^*_{jk}\)` * calcolare le `\(q\)` componenti principali sui dati completi La soluzione del problema di minimo è più complicata rispetto al caso di dati completi, ma è possibile utilizzare il seguente algoritmo iterativo (denominato *hard impute*) --- **Algoritmo iterativo** 1. Per una matrice incompleta di dati `\(X\)`, costruire la matrice `\(\hat{X}\)` con elementi `$$\hat{x}_{ij} = \left\{\begin{array}{cc} x_{ij} & (i,j) \in \mathcal{O} \\ \bar{x}_{j} & (i,j) \notin \mathcal{O} \\ \end{array}\right.$$` dove `\(\bar{x}_{j}\)` è il valore medio della `\(j\)`-sima variabile. 2. Ripetere i passi a.-c. fino a convergenza: a. risolvere `$$\min_{A \in \mathbb{R}^{n\times q}, B \in \mathbb{R}^{p\times q} }\Big\{ \sum_{i=1}^{n}\sum_{j=1}^{p}(\hat{x}_{ij} - \sum_{k=1}^{q} a_{ik} b_{jk} )^2 \Big\}$$` b. Per ogni elemento `\((i,j)\notin\mathcal{O}\)`, `\(\hat{x}_{ij} \leftarrow \sum_{k=1}^{q} a^*_{ik} b^*_{jk}\)` c. Calcolare `$$e = \sum_{(i,j) \in \mathcal{O}}(\hat{x}_{ij} - \sum_{k=1}^{q} a^*_{ik} b^*_{jk} )^2$$` --- ```r Zhat <- Zna zbar <- colMeans(Zna, na.rm = TRUE) Zhat[index.na] <- zbar[inb] ismiss <- is.na(Zna) fit.svd <- function(X, q = 1){ DVS <- svd(X) with(DVS, u[,1:q, drop = FALSE] %*% (d[1:q] * t(v[,1:q, drop = FALSE])) ) } ``` --- ```r cor(Zhat[ismiss], Z[ismiss]) ``` ``` [1] -0.3321775 ``` ```r plot(Zhat[ismiss], Z[ismiss], asp=1) abline(a=0,b=1) ``` 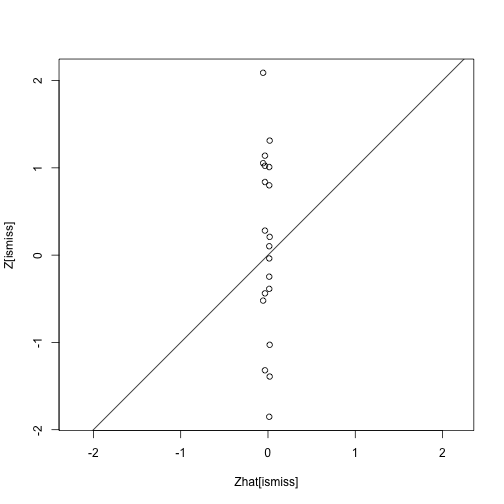<!-- --> --- ```r n_iter <- 10 # numero di iterazioni for (iter in 1:n_iter){ # Step 2.a Zapp <- fit.svd(Zhat, q = 1) # Step 2.b Zhat[ismiss] <- Zapp[ismiss] # Step 2.c e <- mean(((Zna - Zapp)[!ismiss])^2) cat("Iter :", iter, " Errore :", e, "\n") } ``` ``` Iter : 1 Errore : 0.4039168 Iter : 2 Errore : 0.3868899 Iter : 3 Errore : 0.3851522 Iter : 4 Errore : 0.3849471 Iter : 5 Errore : 0.3849151 Iter : 6 Errore : 0.3849085 Iter : 7 Errore : 0.3849069 Iter : 8 Errore : 0.3849065 Iter : 9 Errore : 0.3849064 Iter : 10 Errore : 0.3849064 ``` --- ```r cor(Zhat[ismiss], Z[ismiss]) ``` ``` [1] 0.7694368 ``` ```r plot(Zhat[ismiss], Z[ismiss], asp=1) abline(a=0,b=1) ``` <!-- --> ---  --- # `heptathlon` Caricare il dataset `heptathlon` presente nella libreria `HSAUR` (installarla se non presente nelle librerie disponibili). 1. Esplorare i dati con sintesi grafiche e numeriche appropriate 2. Ricodificare tutte le 7 gare (`hurdles`, `highjump`, `shot`, `run200m`, `longjump`, `javelin`, `run800m`) nella stessa direzione in modo che i valori "grandi" sono indicativi di una prestazione "migliore" 3. Calcolare la matrice di correlazione tra le 7 gare (utilizzando le variabili ricodificate come specificato in 2.). Commentare il risultato. 4. Costruire la matrice di diagrammi di dispersione ed individuare l'osservazione anomala (*outlier*). Rimuovere l'osservazione anomala dai dati e ricalcolare la matrice di correlazione, commentando il risultato. 5. Calcolare le componenti principali e determinarne il numero utilizzando uno o più criteri appropriati. Costruire il *biplot* e interpretare le prime due componenti principali. 6. Calcolare la correlazione (in valore assoluto) tra il vettore dei punteggi della prima componente principale e la variabile `score`, e commentare. <!-- --- --> <!-- # Olivetti `faces` dataset --> <!-- Dieci immagini per ciascuno dei 40 soggetti, raccolte tra il 1992 e il 1994. --> <!-- Per alcuni soggetti, le immagini sono state scattate in momenti diversi, variando l'illuminazione, le espressioni facciali (occhi aperti/chiusi, sorridenti/non sorridenti) e i dettagli del volto (occhiali/senza occhiali). --> <!-- Tutte le immagini sono state scattate su uno sfondo scuro omogeneo con i soggetti in posizione eretta e frontale (con tolleranza per qualche movimento laterale). --> <!-- Il set di dati originale consisteva di `\(92\times 112\)`, mentre la versione che analizzeremo è composta da immagini `\(64\times 64\)`. Ciascuna immagine corrisponde ad una colonna (di lunghezza `\(64\times 64 = 4096\)`) della matrice `\(X\)`. --> <!-- ```{r} --> <!-- rm(list=ls()) --> <!-- url <- "https://raw.githubusercontent.com/aldosolari/AE/master/docs/dati/faces.csv" --> <!-- faces <- read.csv2(url) --> <!-- ``` --> <!-- --- --> <!-- ```{r} --> <!-- face <- matrix(faces[,1], ncol=64) --> <!-- image(face, col=gray(0:255/255), asp=1) --> <!-- ``` --> <!-- --- --> <!-- ```{r} --> <!-- library(dplyr) --> <!-- showFace <- function(x){ --> <!-- x %>% --> <!-- as.numeric() %>% --> <!-- matrix(nrow = 64, byrow = F) %>% --> <!-- apply(2, rev) %>% --> <!-- t %>% --> <!-- image(col=grey(seq(0, 1, length=256)), xaxt="n", yaxt="n") --> <!-- } --> <!-- showFace(t(faces[,1])) --> <!-- ``` --> <!-- --- --> <!-- ```{r} --> <!-- par(mfrow=c(4, 10)) --> <!-- par(mar=c(0.05, 0.05, 0.05, 0.05)) --> <!-- for (i in 1:40) { --> <!-- showFace(t(faces[,i])) --> <!-- } --> <!-- ``` --> <!-- --- --> <!-- ```{r} --> <!-- X <- t(faces[,c(1:2,11:12,21:22,31:32)]) --> <!-- n = nrow(X) --> <!-- p = ncol(X) --> <!-- Xtilde = scale(X, center=TRUE, scale=FALSE) --> <!-- xbar = colMeans(X) --> <!-- meanface <- matrix(xbar, ncol=64) --> <!-- showFace(meanface) --> <!-- ``` --> <!-- --- --> <!-- ```{r} --> <!-- faceSVD <- svd(Xtilde) --> <!-- V = faceSVD$v --> <!-- par(mfrow=c(2, 4)) --> <!-- par(mar=c(0.05, 0.05, 0.05, 0.05)) --> <!-- for (i in 1:8) { --> <!-- showFace(t(V[,i])) --> <!-- } --> <!-- ``` -->